| Codeforces Round 296 (Div. 1) |
|---|
| Закончено |
Леонид работает в молодом и перспективном стартапе, занимающемся расшифровкой генома человека. По долгу службы, ему приходится решать сложные задачи нахождения определённых паттернов в длинных строках, состояющих из букв 'A', 'T', 'G' и 'C'.
Рассмотрим следующую ситуацию. Имеется фрагмент цепочки ДНК человека, записанный в виде строки S. Для анализа фрагмента требуется найти все вхождения строки T в строку S. Однако, дело осложняется тем, что в исходном фрагменте цепочки могли присутствовать незначительные мутации, которые, тем не менее, усложняют задачу поиска фрагмента. Леонид предложил следующий подход решения этой проблемы.
Зафиксируем целое число k ≥ 0 — степень погрешности. Будем говорить, что строка T имеет вхождение в строку S на позиции i (1 ≤ i ≤ |S| - |T| + 1), если после прикладывания строки T в этой позиции, каждому символу строки T соответствует такой же символ в строке S на расстоянии не более k. Более формально, для любого j (1 ≤ j ≤ |T|) должен сущетвовать такой p (1 ≤ p ≤ |S|), что |(i + j - 1) - p| ≤ k и S[p] = T[j].
Например, в соответствии с данным определением, строка "ACAT" входит в строку "AGCAATTCAT" в позициях 2, 3 и 6.
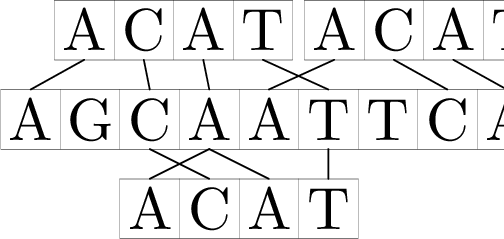
Обратите внимание, что при k = 0 данное определение превращается в обычное определения вхождения строки в строку.
Помогите Леониду — посчитайте, в скольких позициях данная строка T входит в данную строку S при заданной степени погрешности.
В первой строке следуют три целых числа |S|, |T|, k (1 ≤ |T| ≤ |S| ≤ 200 000, 0 ≤ k ≤ 200 000) — длины строк S и T и степень погрешности.
Во второй строке записана строка S.
В третьей строке записана строка T.
Обе строки состоят только из прописных букв 'A', 'T', 'G' и 'C'.
Выведите единственное число — количество вхождений T в S со степенью погрешности k по данному определению.
10 4 1
AGCAATTCAT
ACAT
3
Если вы вдруг разбираетесь в строении генома человека чуть лучше, чем автор задачи, и вы не прониклись оригинальным подходом Леонида, не воспринимайте всё описанное выше всерьёз.
| Название |
|---|




